
中大新聞網訊(通訊員蔡黎)中山大學生命科學學院RNA組學團隊楊建華教授等開發了Pol3Base數據和信息庫,系統鑒定RNA聚合酶III(PolⅢ)轉錄的非編碼RNA(P3RNA),全面解析P3RNA的互作組、表達、進化、表觀轉錄組、疾病變異及調控網絡。
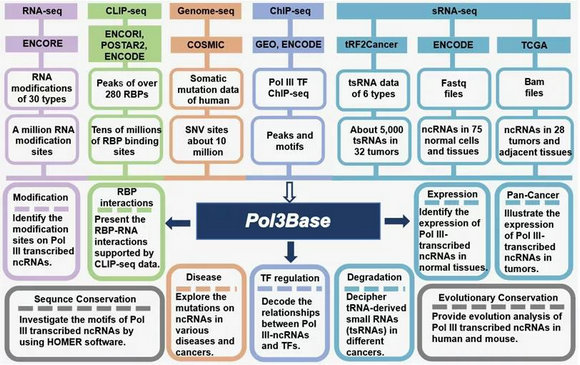
Pol3Base網站大綱。Pol3Base 網站整合了由不同轉錄因子調控的數千個 RNA 聚合酶 III 轉錄的 ncRNAs 轉錄組數據。Pol3Base 的所有結果都存儲在MySQL數據庫,并通過網頁展示。
RNA組學團隊率先開發一系列分析流程去發掘~79000個PolⅢ轉錄的非編碼RNA(P3RNA)與轉錄因子的互作組以及這些P3RNA與240多種RNA結合蛋白之間的相互作用網絡;揭示了70個正常組織和28種不同腫瘤類型中P3RNA的表達譜及9700個表觀RNA修飾位點。此外,通過比較人類和小鼠,發現了大約4000個進化保守的P3RNA;在32個不同的腫瘤組織中鑒定了上萬種 tsRNA。通過分析體細胞突變數據,Pol3Base還提供研究了這些ncRNA的突變圖譜,以幫助揭示它們在多種疾病中的潛在作用。Pol3Base 的訪問網址:http://rna.sysu.edu.cn/pol3base/ 或者http:// biomed.nscc-gz.cn/DB/Pol3Base/。
該研究近日在Nucleic Acids Res雜志上在線發表,楊建華教授、屈良鵠教授、李斌副特聘研究員和鄭凌伶副教授為論文的共同通訊作者。蔡黎、宣佳佳博士生為共同第一作者。
近年來,該團隊建立了國際先進的RNA組學平臺:基于非編碼RNA與其靶標及非編碼RNA與蛋白的互作規律構建了新的罰分矩陣模型,開發了starBase2.0、snoSeeker、RMBase、tsRFun等新一代功能RNA組學技術及在線分析平臺。這些技術在海量的CLIP-seq等測序數據中高精度地發掘出成千上萬的非編碼RNA與靶標、非編碼RNA與蛋白之間的功能互作及網絡。與國際類似方法比較,這些方法顯著地降低了非編碼RNA功能互作網絡預測的假陽性率。研究成果在Nucleic Acids Res等發表系列論文11篇,starBase 2.0 還入選“2014年中國百篇最具影響國際學術論文”,現已被Nature、Cell等雜志他引超過2000次。采用這些技術,該團隊與國際同行合作解析mRNA中m6A修飾生成機制和新的m6A修飾讀碼器等突破性成果已發表在Nature(2019)和Nature Cell Biology(2018)等國際重要雜志上。
論文鏈接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab1033/6423189